无监督学习——DBSCAN
序
与之前提到的凝聚聚类,K均值聚类类似,DBSCAN也是一个非常有用的聚类算法。它的主要优点是它不需要用户先验地设置簇的个数,可以划分具有复杂形状的簇,还可以找出不属于任何簇的点。DBSCAN比凝聚聚类和k均值稍慢,但仍可以扩展到相对较大的数据集。
接下来,我“简单”地介绍一下算法的原理(PS:大概看一下就好)。
算法原理
DBSCAN的全称是具有噪声的基于密度的空间聚类应用。顾名思义,DBSCAN的原理是识别特征空间的“拥挤”区域中的点,在这些区域中许多数据点靠近在一起。这些区域被称为特征空间中的密集区域。DBSCAN背后的思想是,簇形成数据的密集区域,并由相对较空的区域分隔开。
在密集区域内的点被称为核心样本(或核心点),它们的定义如下。DBSCAN有两个参数:min_samples和eps。如果在距一个给定数据点eps的距离内至少有min_samples个数据点,那么这个数据点就是核心样本。DBSCAN将彼此距离小于eps的核心样本放到同一个簇中。
算法首先任意选取一个点,然后找到到这个点的距离小于等于eps的所有的点。如果距起始点的距离在eps之内的数据点个数小于min_samples,那么这个点被标记为噪声,也就是说它不属于任何簇。如果距离在eps之内的数据点个数大于min_samples,则这个点被标记为核心样本,并被分配一个新的簇标签。然后访问该点的所有邻居(在距离eps以内)。如果它们还没有被分配一个簇,那么就将刚刚创建的新的簇标签分配给它们。如果它们是核心样本,那么就依次访问其邻居,以此类推。簇逐渐增大,直到在簇的eps距离内没有更多的核心样本为止。然后选取另一个尚未被访问过的点,并重复相同的过程。
最后,一共有三种类型的点:核心点,与核心点的距离在eps之内的点(叫做边界点)和噪声。如果DBSCAN算法在特定数据集上多次运行,那么核心点的聚类始终相同,同样的点也始终被标记为噪声。但边界点可能与不止一个簇的核心样本相邻。因此,边界点所属的簇依赖于数据点的访问顺序。一般来说只有很少的边界点,这种对访问顺序的轻度依赖并不重要。
接下来,我们对一个数据集two_moons来应用DBSCAN算法。
数据应用
之前,我们对two_moons数据集应用过凝聚聚类和K均值聚类算法,但是聚类结果却都不理想: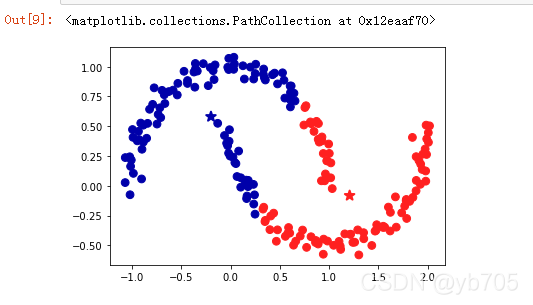
既然如此,我们用DBSCAN算法来试试。
要注意的是,虽然DBSCAN不需要显式地设置簇的个数,但设置eps可以隐式地控制找到的簇的个数。使用StandardScaler或MinMaxScaler对数据进行缩放之后,有时会更容易找到eps的较好取值,因为使用这些缩放技术将确保所有特征具有相似的范围。
(PS:关于数据缩放方面的内容可以看一看这篇文章:数据预处理与缩放,具体的我就不再赘述了。)
所以我们先对原有数据集进行标准化:
1 | |
与凝聚聚类类似,DBSCAN也不允许对新的测试数据进行预测,所以我们将使用fit_predict方法来执行聚类并返回簇标签:
1 | |
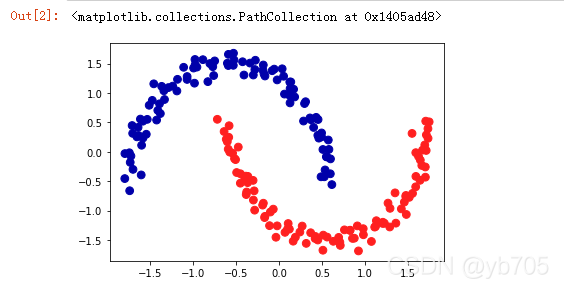
可以看到,利用DBSCAN的默认设置,算法找到了两个半圆形并将其分开。
由于算法找到了我们想要的簇的个数(2个),因此参数设置的效果很好。如果将eps减小到0.2(默认值为0.5),我们将会得到8个簇,这显然太多了。将eps增大到0.7则会导致只有一个簇。
实例应用
现在,我们用另一个数据集来讲解DBSCAN的参数——eps和min_samples。
1.数据来源
胎儿健康分类:https://www.kaggle.com/andrewmvd/fetal-health-classification
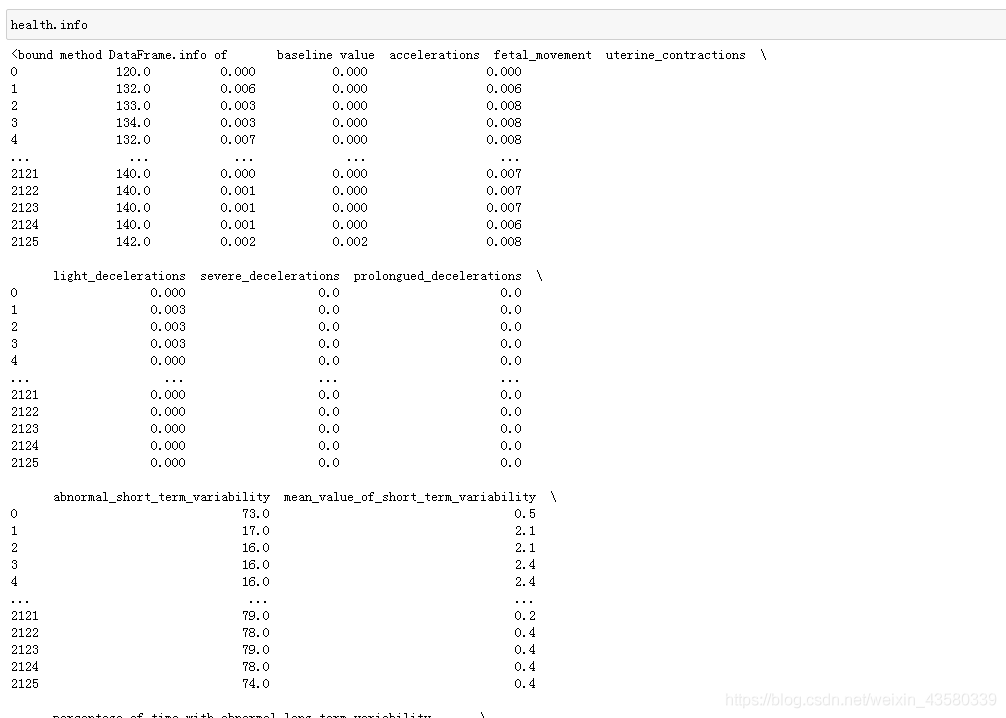
该数据包含胎儿心电图,胎动,子宫收缩等特征值,而我们所需要做的就是通过这些特征值来对胎儿的健康状况(fetal_health)进行分类。

数据集包含从心电图检查中提取的2126条特征记录,然后由三名产科专家将其分为3类,并用数字来代表:1-普通的,2-疑似病理,3-确定病理。
2.导入第三方库并读取文件
1 | |
老规矩,上来先依次导入建模需要的各个模块,并读取文件。
3.对数据集进行缩放以及聚类
1 | |

其中set()方法是用于去重,并查看这一列都有哪些具体元素。
可以看到,在默认设置的情况下,该数据集被DBSCAN分成了6类(其中-1是指噪声)。
接下来,我们尝试不同的参数组合,来具体讲解一下:
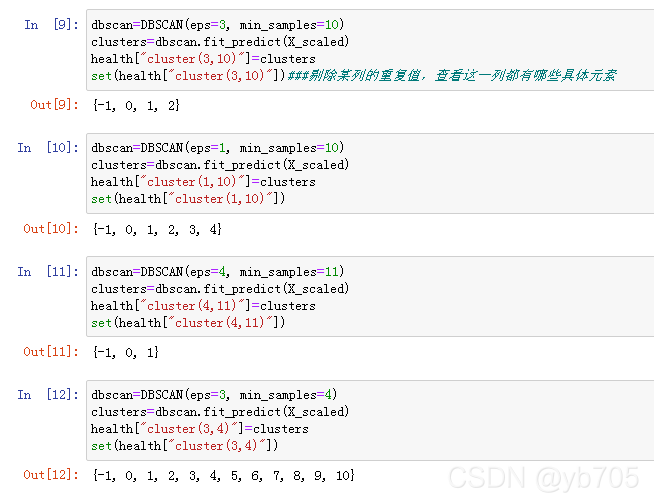
其实参数eps在某种程度上更重要,因为它决定了点与点之间“接近”的含义。将eps设置得非常小,意味着没有点是核心样本,可能会导致所有点都被标记为噪声。将eps设置得非常大,可能会导致所有点形成单个簇。而设置min_samples主要是为了判断稀疏区域内的点被标记为异常值还是形成自己的簇。如果增大min_samples,任何一个包含少于min_samples个样本的簇现在将被标记为噪声。因此,min_samples决定簇的最小尺寸。
总结
在使用DBSCAN时,你需要谨慎处理返回的簇分配。如果使用簇标签对另一个数据进行索引,那么使用-1表示噪声可能会产生意料之外的结果。
个人博客:https://www.yyb705.com/
欢迎大家来我的个人博客逛一逛,里面不仅有技术文,也有系列书籍的内化笔记。
有很多地方做的不是很好,欢迎网友来提出建议,也希望可以遇到些朋友来一起交流讨论。
本博客所有文章除特别声明外,均采用 CC BY-SA 4.0 协议 ,转载请注明出处!